پرونده:Coronavirus. SARS-CoV-2.png
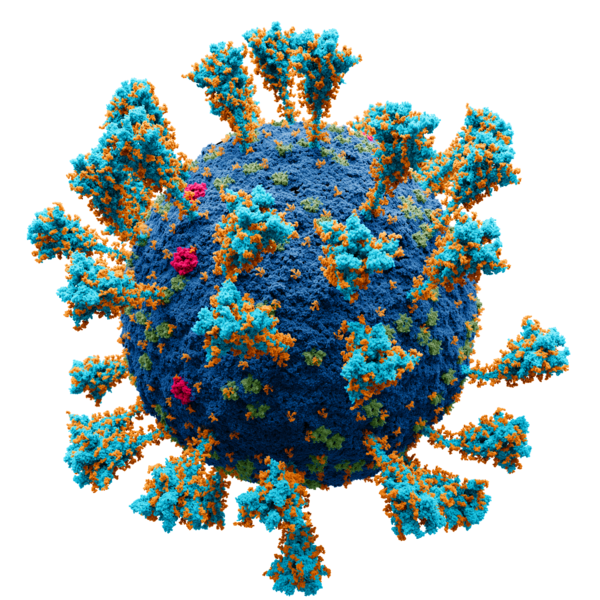
اندازهٔ این پیشنمایش: ۶۰۰ × ۶۰۰ پیکسل. کیفیتهای دیگر: ۲۴۰ × ۲۴۰ پیکسل | ۴۸۰ × ۴۸۰ پیکسل | ۷۶۸ × ۷۶۸ پیکسل | ۱٬۰۲۴ × ۱٬۰۲۴ پیکسل | ۲٬۰۴۸ × ۲٬۰۴۸ پیکسل.
پروندهٔ اصلی (۲٬۰۴۸ × ۲٬۰۴۸ پیکسل، اندازهٔ پرونده: ۴٫۵۴ مگابایت، نوع MIME پرونده: image/png)
تاریخچهٔ پرونده
روی تاریخ/زمانها کلیک کنید تا نسخهٔ مربوط به آن هنگام را ببینید.
| تاریخ/زمان | بندانگشتی | ابعاد | کاربر | توضیح | |
|---|---|---|---|---|---|
| کنونی | ۹ ژانویهٔ ۲۰۲۲، ساعت ۲۲:۱۷ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۴٫۵۴ مگابایت) | Jul059 | Lossless file size reduction |
| ۲۴ سپتامبر ۲۰۲۱، ساعت ۰۳:۵۸ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۴٫۶ مگابایت) | Iketsi | lossless compression | |
| ۱۵ ژوئن ۲۰۲۱، ساعت ۱۶:۰۶ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۵٫۳۴ مگابایت) | AlexeySolodovnikov | fix color bug | |
| ۱۳ ژوئن ۲۰۲۱، ساعت ۱۴:۲۸ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۵٫۳۴ مگابایت) | AlexeySolodovnikov | Мы обновили модель. В роли нашего научного консультанта выступил доктор биологических наук, специалист в области вирусологии, Никитин Н. А. и к.х.н специалист по молекулярному моделированию поверхностных вирусных белков Борисевич С.С. Под их руководством в модель были внесены следующие правки: Изменено количество S-белков с 90 до 38, количество M-белков было увеличено до 1000, а E-белков, как минорных компонентов мембраны, снижено до 15, HE-белок удалён. Также была принята во внимание шарни... | |
| ۱۷ مهٔ ۲۰۲۱، ساعت ۱۱:۰۶ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۱۶٫۰۴ مگابایت) | AlexeySolodovnikov | add alpha | |
| ۴ مهٔ ۲۰۲۱، ساعت ۱۸:۴۱ |  | ۲٬۰۴۸ در ۲٬۰۴۸ (۱۶٫۰۴ مگابایت) | AlexeySolodovnikov | Uploaded own work with UploadWizard |
کاربرد پرونده
صفحههای زیر از این تصویر استفاده میکنند:
کاربرد سراسری پرونده
ویکیهای دیگر زیر از این پرونده استفاده میکنند:
- کاربرد در alt.wikipedia.org
- کاربرد در ar.wikipedia.org
- مراكز السيطرة على الأمراض والوقاية منها
- فيروس كورونا
- مستخدم:Amira Hashem1996/ملعب
- مناطق انتشار جائحة فيروس كورونا حسب الدولة والمنطقة
- عزل ووهان 2020
- قائمة حوادث كراهية الأجانب والعنصرية المرتبطة بجائحة فيروس كورونا
- مستشفى هوو شين شان
- مستشفى لي شين شان
- جائحة فيروس كورونا في العراق
- معهد ووهان لأبحاث الفيروسات
- جائحة فيروس كورونا في إيطاليا
- جائحة فيروس كورونا في الجزائر
- جائحة فيروس كورونا في اليونان
- اللجنة الوطنية للصحة (الصين)
- جائحة فيروس كورونا في الكويت
- جائحة فيروس كورونا في الكاميرون
- المركز الصيني لمكافحة الأمراض والوقاية منها
- جائحة فيروس كورونا في البوسنة والهرسك
- أثر جائحة فيروس كورونا على الحياة الاجتماعية
- مستشفى ووهان المركزي
- جائحة فيروس كورونا في الأردن
- أثر جائحة فيروس كورونا على الرياضة
- جائحة فيروس كورونا في السودان
- جائحة فيروس كورونا في فرنسا
- جائحة فيروس كورونا في إفريقيا
- جائحة فيروس كورونا في جمهورية الكونغو الديمقراطية
- جائحة فيروس كورونا في الغابون
- انهيار فندق شينجيا إكسبريس
- جائحة فيروس كورونا في توغو
- جائحة فيروس كورونا في غينيا
- جائحة فيروس كورونا في رواندا
- جائحة فيروس كورونا في ساحل العاج
- جائحة فيروس كورونا في ناميبيا
- جائحة فيروس كورونا في كينيا
- جائحة فيروس كورونا في مايوت
- جائحة فيروس كورونا في لا ريونيون
- قيود السفر بسبب جائحة فيروس كورونا
- جائحة فيروس كورونا في غينيا الاستوائية
- جائحة فيروس كورونا في جمهورية إفريقيا الوسطى
- جائحة فيروس كورونا في جمهورية الكونغو
- جائحة فيروس كورونا في سيشل
- جائحة فيروس كورونا في ليبيريا
- جائحة فيروس كورونا في الصومال
- جائحة فيروس كورونا في تنزانيا
- جائحة فيروس كورونا في كازاخستان
- جائحة فيروس كورونا في أوروبا
- لقاح كوفيد-19
- جائحة فيروس كورونا في أوقيانوسيا
- جائحة فيروس كورونا في كولومبيا
نمایش استفادههای سراسری از این پرونده.



