ویروس آرانای تکرشتهای با سوی منفی
ویروس RNA تکرشتهای با سوی منفی یا ویروس RNA تکرشتهای سنس منفی (به انگلیسی: Negative-strand RNA virus) که به صورت خلاصه ویروس -ssRNA نوشته میشود، ویروسی است که از سوی منفی و RNA تکرشتهای به عنوان مواد ژنتیکی استفاده میکند. ویروسهای RNA بسته به قطبیت یا سوی شان خود به دو دستهٔ مثبت و منفی تقسیم میشوند. RNA ویروسی منفی کامل کنندهٔ RNA پیامرسان است و باید پیش از ترجمه، توسط RNA پلیمراز به RNA مثبت تبدیل شود. درنتیجه RNA خالص یک ویروس با سوی منفی به خودی خود آلودهکننده نیست بلکه باید به RNA با سوی مثبت تبدیل شود. این ویروسها در گروه ۵ دستهبندی بالتیمور جای دارند.[۳]
| نگارناویریکوتا | |
|---|---|
| |
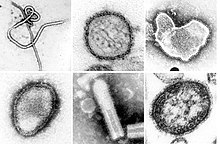
| مونتاژ میکروگرافی الکترونی برخی از ویروسها در شاخه «نگارناویریکوتا». گونهها از چپ به راست، از بالا به پایین: ویروس ابولا زئیر، ویروس سین نومبره، ویروس سنسیشیال تنفسی انسانی ، هندرا هنیپوویروس، یک رابدویروس ناشناس، ویروس سرخک. | |
| طبقهبندی ویروسها | |
| (طبقهبندینشده): | ویروس |
| Realm: | ریبوویریا |
| شاخه: | نگارناویریکوتا |
| در سابتراکسا | |
| مترادف[۱][۲] | |
|
Negative-sense RNA viruses (NSVs) | |
RNA ویروسهای سوی منفی ارتباط نزدیکی با بندپایان دارند و میتوانند بهطور غیررسمی بین آنهایی که برای انتقال به بندپایان متکی هستند و ویروسهایی که از ویروسهای بندپایان منشأ میگیرند اما اکنون میتوانند بدون کمک بندپایان در مهرهداران تکثیر شوند، تقسیم شوند. RNA ویروسهای سوی منفی برجسته بندپایان شامل ویروس تب دره ریفت و ویروس پژمردگی لکهدار گوجهفرنگی هستند. RNA ویروسهای سوی منفی قابل توجه مهرهداران شامل ویروس ابولا، هانتاویروسها، ویروسهای آنفولانزا، ویروس تب لاسا و ویروس هاری هستند.
همانندسازی ویرایش
ویروس ssRNA با سوی منفی برای تبدیل به RNA با سوی مثبت به RNA پلیمراز نیاز دارد. RNA با سوی مثبت به عنوان RNA پیامرسان ویروسی عمل میکند که خود برای تولید مواد ویریون جدید، به پروتئین ترجمه میشود. با ویریونهای تازه تولید شده، مولکولهای RNA با سوی منفی بیشتری تولید میشود. گامهای همانندسازی ویریون به قرار زیر است:[۴][۵][۶]
- یک ویریون وارد سلول میزبان میشود و RNA منفی خود را در سیتوپلاسم آزاد میکند.
- ویروس از کپی دقیق RNA خود که RNA پلیمراز وابسته به RNA نام دارد (به صورت خلاصه RdRp) استفاده میکند تا یک الگوی RNA مثبت از جفتباز تکمیلی بسازد.
- کپسیدهای ویروسی جدید با پروتئین خاصی سرهم میشود. رشتههای منفی RNA با کپسیدها و RdRp ویروسی ترکیب میشود تا ویریونهای جدید RNA منفی بسازد.
- پس از سرهم شدن و تطابق نوکلئوکپسیدها، ویریونهای جدید از سلول خارج میشوند و غشای سلولی دیگر سلولها را نابود میکنند تا آلودگی را گسترش دهند.
بزرگی ژنوم یک ویروس RNA منفی میان 10kb تا 30kb است (هر bp برابر با ۳۴۰ پیکومتر است.[۷])
همانندسازی و رونویسی ویرایش
همانندسازی ژنوم RNA ویروسهای دورشتهای توسط RDRV اجرا میشود، که همانندسازی را با اتصال به یک توالی رهبر در انتهای ۳' (معمولاً «سه انتهای اصلی» تلفظ میشود) ژنوم آغاز میکند. سپس RDRV از ژنوم سوی منفی به عنوان الگویی برای سنتز آنتی ژنوم با سوی مثبت استفاده میکند. هنگام تکثیر آنتی ژنوم، RDRV ابتدا به دنباله تریلر در انتهای ۳' آنتی ژنوم متصل میشود. پس از آن، RDRV تمام سیگنالهای رونویسی روی آنتی ژنوم را نادیده میگیرد و یک کپی از ژنوم را در حالی که از آنتی ژنوم به عنوان الگو استفاده میکند سنتز میکند.[۸] همانندسازی زمانی انجام میشود که ژنوم داخل نوکلئوکپسید است و RDRV کپسید را آشکار میکند و در طول همانندسازی در طول ژنوم جابهجا میشود. همانطور که توالیهای نوکلئوتیدی جدید توسط RDRV سنتز میشوند، پروتئینهای کپسید مونتاژ میشوند و آر ان ای ویروسی تازه تکثیر شده را محصور میکنند.
مورفولوژی ویرایش
RNA ویروسهای سوی منفی حاوی یک کمپلکس ریبونوکلئوپروتئینی متشکل از ژنوم و یک RDRV متصل به هر بخش از ژنوم است که توسط یک کپسید احاطه شدهاست.[۹] کپسید از پروتئینهایی تشکیل شدهاست که ساختار چین خورده آنها شامل پنج مارپیچ آلفا در لوب پایانه-N (موتیف 5-H) و سه مارپیچ آلفا در لوب پایانه-C (موتیف 3-H) است. در داخل کپسید، ژنوم بین این دو نقوش قرار گرفتهاست. به استثنای خانواده آسپیویریده، RNA ویروسهای سوی منفی حاوی یک پوشش ویروسی خارجی هستند، نوعی غشای لیپیدی که کپسید را احاطه کردهاست. شکل ذره ویروس که ویریون نامیده میشود، RNA ویروسهای سوی منفی متفاوت است و ممکن است رشتهای، پلئومورفیک، کروی یا لولهای باشد.
فرگشت ویرایش
تقسیمبندی ژنوم یک ویژگی برجسته در میان بسیاری از ویروسهای -اس اس آر ان ای است، و ویروسهای -اس اس آر ان ای از داشتن ژنوم با یک بخش، عادی برای اعضای راسته مونونگاویرالس، تا ژنومهایی با ده بخش، مانند ویروس تیلاپیا تیلاپین، متغیر است.[۱۰][۱۱] هیچ روند مشخصی در طول زمان وجود ندارد که تعداد بخشها را تعیین کند، و به نظر میرسد تقسیمبندی ژنوم در میان RNA ویروسهای سوی منفی یک ویژگی انعطافپذیر باشد زیرا بهطور مستقل در موارد متعدد فرگشت یافتهاست. اکثر اعضای زیر شاخه هاپلوویریکوتینا غیرقطعی هستند، در حالی که تقسیمبندی در Polyploviricotina جهانی است.[۱۰]
فیلوژنتیک ویرایش
تجزیه و تحلیل فیلوژنتیکی بر اساس RDRV نشان میدهد که RNA ویروسهای سوی منفی از یک نیای مشترک منشأ میگیرند و احتمالاً یک کلاد خواهر از رئوویروسها هستند که ویروسهای دی اس آر ان ای هستند. در داخل شاخه، دو شاخه واضح وجود دارد که به دو زیر شاخه اختصاص داده میشوند، بر این اساس که آیا RDRV یک کلاهک را روی ام آر ان ای ویروسی سنتز میکند یا یک کلاهک را از ام آر ان ای میزبان ربوده و آن کلاه را به ام آر ان ای ویروسی متصل میکند.[۱۲]
در این شاخه، ویروسهای -اس اس آر ان ای که بندپایان را آلوده میکنند، ظاهراً پایه و اجداد سایر ویروسهای -اس اس آر ان ای هستند. بندپایان اغلب در گروههای بزرگ با هم زندگی میکنند، که امکان انتقال آسان ویروسها را فراهم میکند. با گذشت زمان، این باعث شدهاست که RNA ویروسهای بندپایان سطح بالایی از تنوع را به دست آورند. در حالی که بندپایان میزبان مقادیر زیادی ویروس هستند، در مورد میزان انتقال بین گونهای ویروسهای بندپایان RNA ویروسهای سوی منفی در بین بندپایان اختلاف نظر وجود دارد.[۱۳][۱۰]
RNA ویروسهای سوی منفی گیاهان و مهرهداران از نظر ژنتیکی با ویروسهای آلوده به بندپایان مرتبط هستند. علاوه بر این، بیشتر RNA ویروسهای سوی منفی خارج از بندپایان در گونههایی یافت میشوند که با بندپایان تعامل دارند؛ بنابراین بندپایان هم به عنوان میزبان کلیدی و هم به عنوان ناقل انتقال RNA ویروسهای سوی منفی عمل میکنند. از نظر انتقال، RNA ویروسهای سوی منفی غیر بندپایان را میتوان بین ویروسهایی که برای انتقال به بندپایان متکی هستند و ویروسهایی که میتوانند بدون کمک بندپایان در بین مهرهداران گردش کنند، تمایز قائل شد. گروه دوم احتمالاً از اولی سرچشمه گرفتهاند و با انتقال فقط مهره داران سازگار شدهاند.[۱۰]
تبارشناسی ویرایش
تاکنون یک شاخه، دو زیرشاخه، هشت راسته و بیست و یک خانواده برای این گروه شناسایی شدهاست.[۱۴] البته مواردی وجود دارند که هنوز در هیچ دسته ای جایگذاری نشدهاند.[۱۵]
بیماری ویرایش
RNA ویروسهای سوی منفی باعث بسیاری از بیماریهای شناخته شده میشوند. بسیاری از اینها توسط بندپایان منتقل میشوند، از جمله ویروس تب دره ریفت و ویروس پژمردگی خالدار گوجه فرنگی.[۱۶][۱۷] در میان مهره داران، خفاشها و جوندگان ناقل رایج بسیاری از ویروسها هستند، از جمله ویروس ابولا و ویروس هاری که توسط خفاشها و سایر مهرهداران منتقل میشوند،[۱۸][۱۹] و ویروس تب لاسا و هانتاویروسها که توسط جوندگان منتقل میشوند.[۲۰][۲۱] ویروس آنفولانزا در بین پرندگان و پستانداران رایج است.[۲۲] RNA ویروسهای سوی منفی خاص انسان شامل ویروس سرخک و ویروس اوریون هستند.[۲۳][۲۴]
تاریخچه ویرایش
بسیاری از بیماریهای ناشی از RNA ویروسهای سوی منفی در طول تاریخ شناخته شدهاند، از جمله عفونت هانتاویروس، سرخک و هاری.[۲۵][۲۶][۲۷] در تاریخ مدرن، برخی مانند ابولا و آنفولانزا باعث شیوع بیماریهای مرگبار شدهاند.[۲۸][۲۹] ویروس استوماتیت تاولی که برای اولین بار در سال ۱۹۲۵ جدا شد و یکی از اولین ویروسهای حیوانی مورد مطالعه قرار گرفت، زیرا میتوان آن را در کشتهای سلولی به خوبی مطالعه کرد، به عنوان یک RNA ویروسهای سوی منفی شناسایی شد که در آن زمان منحصربهفرد بود زیرا سایر RNA ویروسها که قبلاً شناسایی شده بودند. کشف شده سوی مثبت بودند.[۳۰][۳۱] در اوایل قرن بیست و یکم، بیماری گاوی که توسط ویروس طاعون گاوی ایجاد میشود، پس از آبله که توسط یک دیانای ویروس ایجاد میشود، به دومین بیماری ریشهکنشده تبدیل شد.[۳۲]
در قرن بیست و یکم، متاژنومیکس ویروسی برای شناسایی ویروسهای موجود در محیط رایج شدهاست. برای RNA ویروسهای سوی منفی، این امکان شناسایی تعداد زیادی از بیمهرگان، و بهویژه بندپایان، ویروسها را فراهم کرد که به ارائه بینشی از تاریخچه تکاملی RNA ویروسهای سوی منفی کمک کرد. بر اساس تجزیه و تحلیل فیلوژنتیکی RDRV که نشان میدهد RNA ویروسهای سوی منفی از یک جد مشترک، Negarnaviricota و دو زیر شاخه آن در سال ۲۰۱۸ تأسیس شدهاند، و در قلمرو تازه تأسیس آن زمان ریبوویریا قرار گرفتهاند.[۳۳]
میزبانان ویرایش
ویروسهای خانوادهٔ آرناویریدا، پارامیکسوویریدا و نیوموویریدا میتوانند مهره داران را بیمار کنند. ویروسهای خانوادهٔ بونیاویریدا و رابدوویریدا میتوانند مهره داران، بندپایان و گیاهان را بیمار کنند. ویروسهای سردهٔ تنوئی ویروس تنها گیاهان را بیمار میکنند. از جمله ویروسهای آلوده کنندهٔ انسان میتوان به ویروس ماربورگ، ابولا، هاری و … اشاره کرد.
منابع ویرایش
- ↑ Wolf Y, Krupovic M, Zhang YZ, Maes P, Dolji V, Koonin EV (21 August 2017). "Megataxonomy of negative-sense RNA viruses" (docx). International Committee on Taxonomy of Viruses (ICTV) (به انگلیسی). Retrieved 6 August 2020.
- ↑ Luo M, Terrel JR, Mcmanus SA (30 July 2020). "Nucleocapsid Structure of Negative Strand RNA Virus". Viruses. 12 (8): 835. doi:10.3390/v12080835. PMC 7472042. PMID 32751700.
- ↑ Baltimore, D (1971). "Expression of animal virus genomes". Bacteriological Reviews. 35 (3): 235–241. doi:10.1128/MMBR.35.3.235-241.1971. PMC 378387. PMID 4329869.
- ↑ Samuel, Charles E (2005). "Virus-Host Interaction Minireview Series: Human Immunodeficiency Virus, Hepatitis C Virus, and Influenza Virus". The Journal of Biological Chemistry. 281 (13): 8305–7. doi:10.1074/jbc.R500032200. PMID 16407181.
- ↑ Hofkin, Bruce (2010). Living in a Microbial World. Garland Science. ISBN 978-0-8153-4175-8.
- ↑ Rybicki, Ed. "Viral Replication Strategy in Negative-Sense Single-Stranded RNA Viruses". ASM Microbe Library. American Society for Microbiology. Archived from the original on 23 October 2014. Retrieved 14 April 2015.
- ↑ Alberts B, Johnson A, Lewis J, Morgan D, Raff M, Roberts K, Walter P (December 2014). Molecular Biology of the Cell (6th ed.). New York/Abingdon: Garland Science, Taylor & Francis Group. p. 177. ISBN 978-0-8153-4432-2.
- ↑ "Negative stranded RNA virus replication". ViralZone. Swiss Institute of Bioinformatics. Retrieved 6 August 2020.
- ↑ Zhou H, Sun Y, Guo Y, Lou Z (September 2013). "Structural perspective on the formation of ribonucleoprotein complex in negative-sense single-stranded RNA viruses". Trends Microbiol. 21 (9): 475–484. doi:10.1016/j.tim.2013.07.006. PMID 23953596.
- ↑ ۱۰٫۰ ۱۰٫۱ ۱۰٫۲ ۱۰٫۳ Li CX, Shi M, Tian JH, Lin XD, Kang YJ, Chen LJ, Qin XC, Xu J, Holmes EC, Zhang YZ (29 January 2015). "Unprecedented genomic diversity of RNA viruses in arthropods reveals the ancestry of negative-sense RNA viruses". eLife. 4 (4): e05378. doi:10.7554/eLife.05378. PMC 4384744. PMID 25633976. Retrieved 6 August 2020.
- ↑ Bacharach E, Mishra N, Briese T, Zody MC, Kembou Tsofack JE, Zamostiano R, Berkowitz A, Ng J, Nitido A, Corvelo A, Toussaint NC, Abel Nielsen SC, Hornig M, Del Pozo J, Bloom T, Ferguson H, Eldar A, Lipkin WI (5 April 2016). "Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia". mBio. 7 (2): e00431-16. doi:10.1128/mBio.00431-16. PMC 4959514. PMID 27048802.
- ↑ Wolf YI, Kazlauskas D, Iranzo J, Lucia-Sanz A, Kuhn JH, Krupovic M, Dolja VV, Kooning EV (27 November 2018). "Origins and Evolution of the Global RNA Virome". mBio. 9 (6): e02329-18. doi:10.1128/mBio.02329-18. PMC 6282212. PMID 30482837.
- ↑ Käfer S, Paraskevopoulou S, Zirkel F, Wieseke N, Donath A, Petersen M, Jones TC, Liu S, Zhou X, Middendorf M, Junglen S, Misof B, Drosten C (12 December 2019). "Re-assessing the diversity of negative strand RNA viruses in insects". PLOS Pathog. 15 (12): e1008224. doi:10.1371/journal.ppat.1008224. PMC 6932829. PMID 31830128.
- ↑ Cann, Alan (2011). Principles of Molecular Virology. Academic Press. ISBN 978-0-12-384939-7.
- ↑ Klein, Donald W.; Prescott, Lansing M.; Harley, John (1993). Microbiology. Dubuque, Iowa: Wm. C. Brown. ISBN 978-0-697-01372-9.
- ↑ Hartman A (June 2017). "Rift Valley Fever". Clin Lab Med. 37 (2): 285–301. doi:10.1016/j.cll.2017.01.004. PMC 5458783. PMID 28457351.
- ↑ Scholthof KB, Adkins S, Czosnek H, Palukaitis P, Jacquot E, Hohn T, Hohn B, Saunders K, Candresse T, Ahlquist P, Hemenway C, Foster GD (December 2011). "Top 10 plant viruses in molecular plant pathology". Mol Plant Pathol. 12 (9): 938–954. doi:10.1111/j.1364-3703.2011.00752.x. PMC 6640423. PMID 22017770.
- ↑ Muñoz-Fontela C, McElroy AK (2017). "Ebola Virus Disease in Humans: Pathophysiology and Immunity". Curr Top Microbiol Immunol. Current Topics in Microbiology and Immunology. 411: 141–169. doi:10.1007/82_2017_11. ISBN 978-3-319-68946-3. PMC 7122202. PMID 28653186.
- ↑ Fisher CR, Streicker DG, Schnell MJ (April 2018). "The spread and evolution of rabies virus: conquering new frontiers". Nat Rev Microbiol. 16 (4): 241–255. doi:10.1038/nrmicro.2018.11. PMC 6899062. PMID 29479072.
- ↑ Yun NE, Walker DH (9 October 2012). "Pathogenesis of Lassa fever". Viruses. 4 (10): 2031–2048. doi:10.3390/v4102031. PMC 3497040. PMID 23202452.
- ↑ Avsic-Zupanc T, Saksida A, Korva M (April 2019). "Hantavirus infections". Clin Microbiol Infect. 21S: e6–e16. doi:10.1111/1469-0691.12291. PMID 24750436. Retrieved 6 August 2020.
- ↑ Borkenhagen LK, Salman MD, Ma MJ, Gray GC (November 2019). "Animal influenza virus infections in humans: A commentary". Int J Infect Dis. 88: 113–119. doi:10.1016/j.ijid.2019.08.002. PMID 31401200. Retrieved 6 August 2020.
- ↑ "Transmission of Measles". cdc.gov. Centers for Disease Control and Prevention (CDC). 5 February 2018. Retrieved 6 August 2020.
- ↑ Rubin S, Eckhaus M, Rennick LJ, Bamford CG, Duprex WP (January 2015). "Molecular biology, pathogenesis and pathology of mumps virus". J Pathol. 235 (2): 242–252. doi:10.1002/path.4445. PMC 4268314. PMID 25229387.
- ↑ Jiang H, Zheng X, Wang L, Du H, Wang P, Bai X (2017). "Hantavirus infection: a global zoonotic challenge". Virol Sin. 32 (1): 32–43. doi:10.1007/s12250-016-3899-x. PMC 6598904. PMID 28120221.
- ↑ "Measles history". cdc.gov. Centers for Disease Control and Prevention (CDC). 5 February 2018. Retrieved 6 August 2020.
- ↑ Velasco-Villa A, Mauldin MR, Shi M, Escobar LE, Gallardo-Romero NF, Damon I, Olson VA, Streicker DG, Emerson G (October 2017). "The history of rabies in the Western Hemisphere". Antiviral Res. 146: 221–232. doi:10.1016/j.antiviral.2017.03.013. PMC 5620125. PMID 28365457.
- ↑ Zawilinska B, Kosz-Vnenchak M (2014). "General introduction into the Ebola virus biology and disease" (PDF). Folia Med Cracov. 54 (3): 57–65. PMID 25694096. Retrieved 6 August 2020.
- ↑ Krammer F, Smith G, Fouchier R, Peiris M, Kedzierska K, Doherty PC, Palese P, Shaw ML, Treanor J, Webster RG, García-Sastre A (28 June 2018). "Influenza". Nat Rev Dis Primers. 4 (1): 3. doi:10.1038/s41572-018-0002-y. PMC 7097467. PMID 29955068.
- ↑ "Vesicular stomatitis virus" (PDF). Swine Health Information Center. Center for Food Security and Public Health, College of Veterinary Medicine, Iowa State University. November 2015. Retrieved 6 August 2020.
- ↑ Kolakofsky D (April 2015). "A short biased history of RNA viruses". RNA. 21 (4): 667–669. doi:10.1261/rna.049916.115. PMC 4371325. PMID 25780183. Retrieved 6 August 2020.
- ↑ Greenwood B (12 May 2014). "The contribution of vaccination to global health: past, present and future". Philos Trans R Soc Lond B Biol Sci. 369 (1645): 20130433. doi:10.1098/rstb.2013.0433. PMC 4024226. PMID 24821919.
- ↑ "ICTV Taxonomy history: Negarnaviricota". Internatinal Committee on Taxonomy of Viruses (ICTV). Retrieved 6 August 2020.